Fr, 10.05.2013 - 04:20 — Peter Palese
 Peter Palese, weltweit einer der renommiertesten Virologen, hat entscheidend zur genetischen Analyse und Aufklärung der Funktion von Genen und Genprodukten der Influenzaviren beigetragen. Die Bausteine dieser Viren verändern sich laufend und führen rasch zur Resistenz gegen aktuell wirksame Grippeimpfungen und Medikamente. Um lebensbedrohende Epidemien durch neue, hochinfektiöse Virenstämme abwehren zu können, erscheint die Entwicklung universell gegen unterschiedliche Stämme einsetzbarer, langfristig – möglicherweise sogar lebenslang – wirksamer Impfstoffe vordringlich. Der folgende Essay ist eine aus Vorträgen und Publikationen Peter Paleses zusammengegestellte und von ihm autorisierte Fassung.
Peter Palese, weltweit einer der renommiertesten Virologen, hat entscheidend zur genetischen Analyse und Aufklärung der Funktion von Genen und Genprodukten der Influenzaviren beigetragen. Die Bausteine dieser Viren verändern sich laufend und führen rasch zur Resistenz gegen aktuell wirksame Grippeimpfungen und Medikamente. Um lebensbedrohende Epidemien durch neue, hochinfektiöse Virenstämme abwehren zu können, erscheint die Entwicklung universell gegen unterschiedliche Stämme einsetzbarer, langfristig – möglicherweise sogar lebenslang – wirksamer Impfstoffe vordringlich. Der folgende Essay ist eine aus Vorträgen und Publikationen Peter Paleses zusammengegestellte und von ihm autorisierte Fassung.
Virusgrippe – ein enormes Gesundheitsproblem
Jährlich erkranken Millionen Menschen an der Virusgrippe, der Influenza. Die Folgen können fatal sein, vor allem für Patienten, die dem Erreger keine ausreichende körperliche Abwehr entgegensetzen können. Weltweit sterben jährlich im Schnitt bis zu 500 000 Menschen an der Virusgrippe (nach den Daten von CDC WHO Am. Lung Assoc. sind allein in den US jährlich rund 37 000 Todesfälle und mehr als 200 000 Spitalsaufenthalte auf Virusgrippe zurückzuführen, bei geschätzten Kosten von 37,5 Milliarden $)
Im Falle verheerender Epidemien – wie der „Spanischen Grippe“ im Jahre 1918 – können es auch hundert mal so viele sein (Abbildung 1).
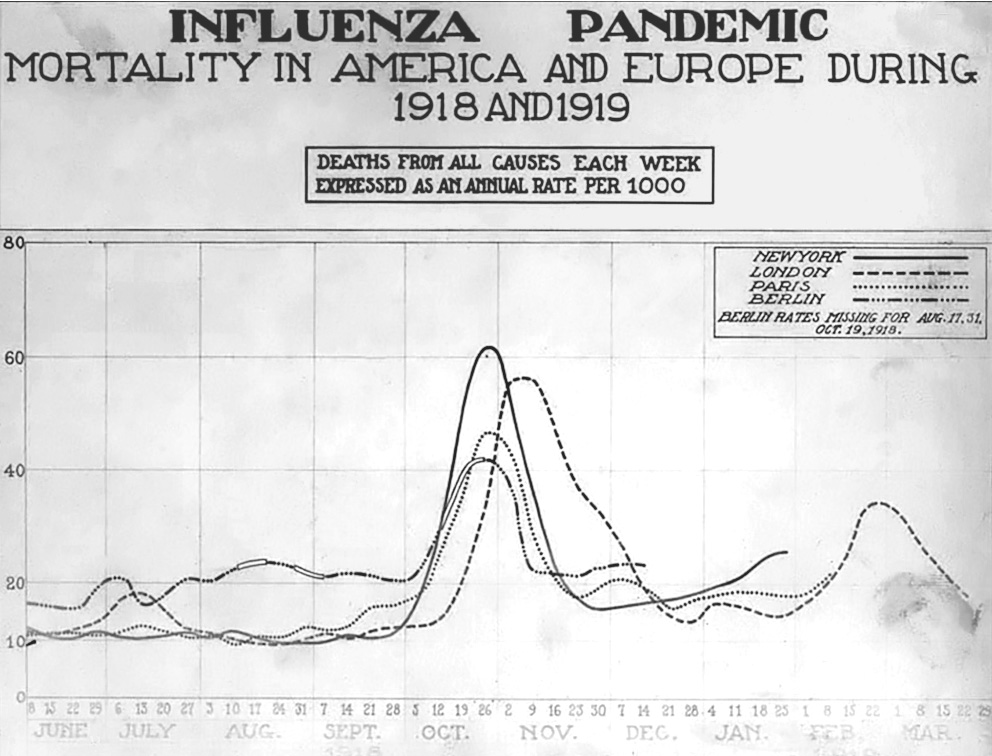 Abbildung 1. Die “Spanische Grippe” von 1918. An dieser Pandemie (= globalen Epidemie) erkrankten rund 30% der Weltbevölkerung, 50 bis 100 Millionen Menschen starben. (Abbildung: US National Museum of Health and Medicine; Wikimedia)
Abbildung 1. Die “Spanische Grippe” von 1918. An dieser Pandemie (= globalen Epidemie) erkrankten rund 30% der Weltbevölkerung, 50 bis 100 Millionen Menschen starben. (Abbildung: US National Museum of Health and Medicine; Wikimedia)
Durch Influenza-Viren verursachte Grippe Epidemien stellen nach wie vor ein enormes Gesundheitsproblem dar. Die Bausteine der Viren mutieren laufend: Viren werden gegen heute noch wirksame Medikamente schnell resistent, ebenso gegen momentan angewandte Impfstoffe. Die Impfstoffe müssen daher jährlich, entsprechend den Empfehlungen der WHO [1], speziell den aktuellen (prognostizierten) Erregertypen angepaßt, Grippe-Impfungen im jährlichen Abstand vorgenommen werden. Diese jährliche Prozedur erweist sich als kostspielig, die Einführung der Impfstoffe als aufwendig und zeitraubend. Aber auch die kontinuierlich erfolgende Anpassung kann einem plötzlich auftretenden, in unserer globalisierten Welt sich rasch verbreitenden, neuen Erregertyp nachhinken.
Das Verstehen der molekularen Grundlagen der Virus-Bausteine und ihrer Funktion, d.h. die Kenntnis darüber, wie sich Viren vermehren und Wirtszellen infizieren, erlaubt erstmals die Entwicklung universell, gegen unterschiedliche Stämme von Influenzaviren wirksamer Impfstoffe in Angriff zu nehmen.
Das molekulare Make-up der Influenza Viren
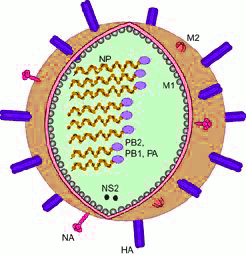
Influenza Viren sind RNA-Viren, d.h. ihre genetische Information liegt in der Ribonukleinsäure – RNA – gespeichert vor und zwar in acht RNA-Segmenten, die je nach Virus Stamm fuer 10-12 Genprodukte kodieren.
Zwei dieser Genprodukte, die Proteine Hemagglutinin und Neuraminidase bilden an der Oberfläche des Virus einen dichten Rasen von „Spikes“ – diese sind das, was bei einer Infektion unser Immunsystem zu sehen bekommt (Abbildung 2). Hemagglutinin bewirkt das Andocken des Virus an der Wirtszelle und seine Aufnahme ins Zellinnere, das Enzym Neuraminidase die Freisetzung der in der Wirtszelle vervielfältigten Viruspartikel und damit deren Verbreitung innerhalb und außerhalb des befallenen Organismus.
Verglichen mit der infizierten Zelle (Durchmesser rund 10 – 20 Mikrometer) ist das Virus sehr klein (Durchmesser rund 100 Nanometer). Innerhalb von 8 Stunden kann es aber hunderttausende neue Partikel bilden, die „riesige“ Wirtszelle Zelle töten und neue Zellen infizieren.
Spurensuche – Influenzaviren im letzten Jahrhundert
Infektionen des Menschen mit Influenzaviren treten in der nördlichen Hemisphäre vorwiegend von November bis März auf und werden vor allem durch das Influenza Virus der Gattung A verursacht. Auch eine Vielzahl an Tierarten – Vögel, Schweine, Pferde, Hunde, Meerestiere, etc. – ist von Influenza A Infektionen betroffen. Influenza A Viren (oder auch einzelne ihrer Gene, z.B. bei „Schweinegrippe“, siehe unten) können dabei auch von einer Spezies auf andere übertragen werden. Infektionen mit Influenza B Virus sind auf den Menschen beschränkt, seltener als Influenza A Infektionen und haben meist einen milderen Verlauf.
Auf Grund einer hohen Fehlerrate während des Kopiervorgangs (Replikation) der Influenza-Gene entstehen in diesen laufend Punktmutationen, welche ihrerseits zur Mutation einzelner Aminosäuren in den von ihnen kodierten Proteinen führen. Im Falle der immunitätsbildenden Oberflächenproteine, insbesondere im Fall von Hemagglutinin, kann die veränderte Aminosäuresequenz dazu führen, daß es von vormals wirksamen Antikörpern nicht mehr erkannt wird, daß das Virus so der Immunabwehr des Wirtsorganismus entkommen kann. Ein sogenannter ›Antigendrift‹ hat stattgefunden. Die serologische Identifizierung von Influenza A erfolgt auf Grund der Oberflächen-Proteine Hemagglutinin (H ) und Neuraminidase (N). Bis jetzt wurden 17 H-Subtypen (H1 – H17) und 10 N-Subtypen (N1 – N9) klassifiziert, die zu unterschiedlichen Kombinationen führen können (H1N1, H2N2,…). Von besonderer Wichtigkeit erscheint die Frage, welche Veränderungen zu einer erhöhten Pathogenität, zu einer länder- und kontinentübergreifenden Ansteckungsgefahr – einer Pandemie – führen können, mit katastrophalen Folgen wie im Fall der „Spanischen Grippe“ im Jahre 1918.
Um die Frage beantworten zu können, was die Erreger der „Spanischen Grippe“ von anderen Epidemien-auslösenden Influenzaviren unterschied, wurde das heute nicht mehr vorhandene 1918-Virus rekonstruiert: Als Ausgangsmaterial dienten Proben aus den Lungen damals verstorbener Soldaten, aus denen mit molekularbiologischen Methoden die viralen Gene isoliert, sequenziert und daraus das damalige Influenzavirus rekonstruiert werden konnte. Mit dem wiederhergestellten Virus konnten nicht nur Fragen hinsichtlich der durch einzelne Gene vermittelten Pathogenität geklärt werden, das Virus dient seitdem auch als exzellenter Standard für das Design von Infektionsmodellen.
Das 1918-Virus gehörte zum H1 Subtyp, dieses Virus wurde 1957 abgelöst durch einen H2 Subtyp („Asiatische Grippe“). Es folgten 1968 ein neuer Subtyp H3 („Hongkong-Grippe“), welcher heute noch vorhanden ist und 1977 ein anderes H1-Virus („Russische Grippe“), welches dem Virus aus den 50er Jahren sehr ähnlich ist. Ein neues Virus des H1N1-Subtyps trat im März 2009 auf („Schweine“- oder „Mexiko“-Grippe), welches eine Pandemie auslöste (bis Juni 2009 bestätigten 105 Länder insgesamt 59814 Fälle und 263 Tote mit dem neuem H1N1-Virus).
Damit koexistieren drei unterschiedliche humanpathogene Virentypen: die H1- und H3-Subtypen und zusätzlich das Influenza B-Virus. Das erklärt warum aktuelle Grippeimpfstoffe dementsprechend drei unterschiedliche Komponenten aufweisen müssen. Da in jüngster Zeit nun zwei unterschiedliche B-Virusstämme zirkulieren, wird die Vakzine der 2013/2014 Saison aus 4 Komponenten bestehen und zusätzlich zum H1 und H3 Hemagglutinin 2 unterschiedliche B-Virus Hemagglutinine enthalten.
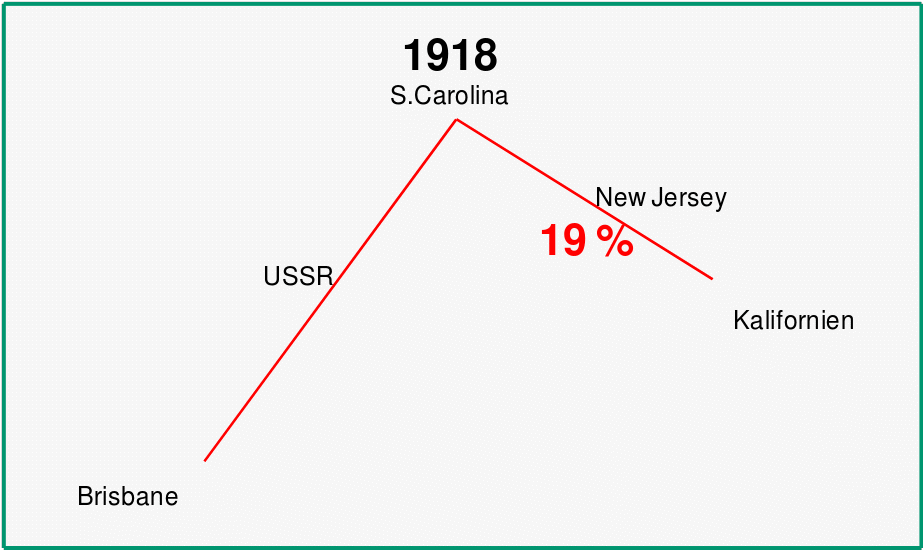
Ein „historischer“ Überblick über die Influenza A Subtypen in der humanen Bevölkerung ist in Abbildung 3 gegeben. 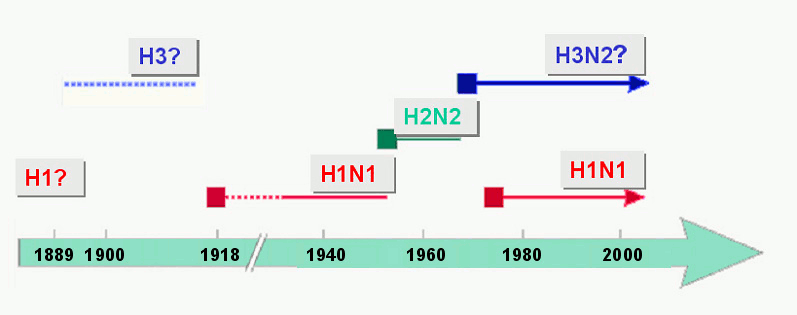
Abbildung 3. Prävalenz von Influenza A Subtypen im vergangenen Jahrhundert
Die 2009 Pandemie (Schweinegrippe)
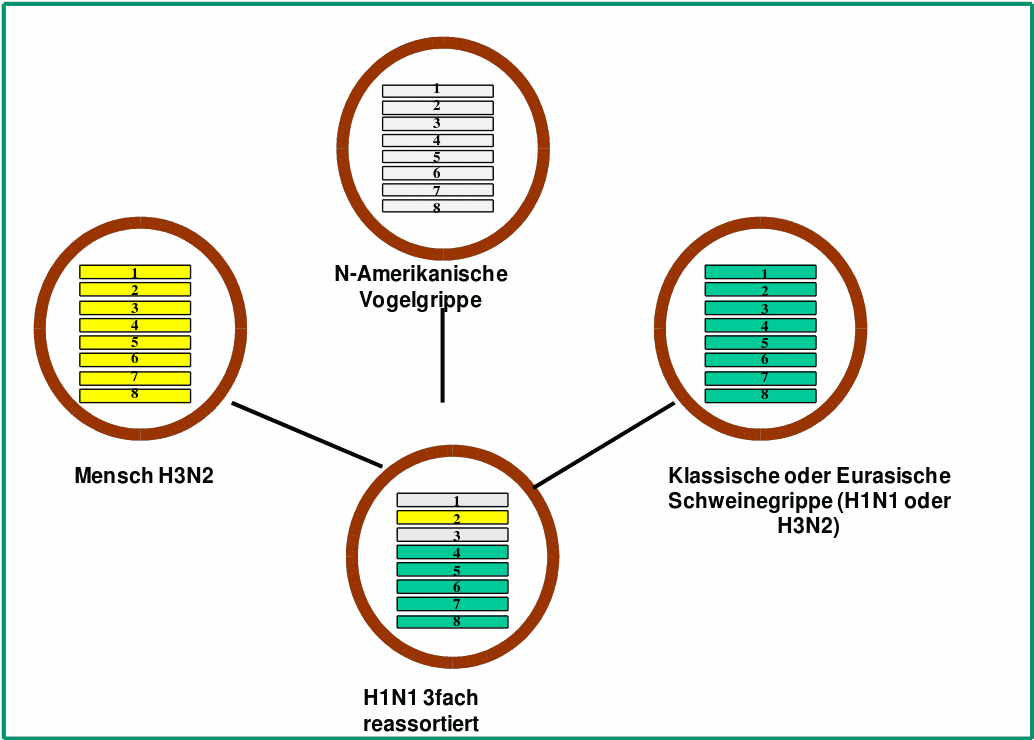
Der zuletzt 2009 aufgetretene H1N1-Subtyp ist in mehrfacher Hinsicht ungewöhnlich. Abgesehen von einer verspäteten, sich über das Frühjahr ausbreitenden Pandemie, stammen die 8 Segmente des Minichromosoms aus verschiedenen Influenza-Subtypen: sie sind durch Neukombination – Reassortment – von 5 Gensegmenten der klassischen und Eurasischen Schweinegrippe, 2 Gensegmenten der Nordamerikanischen Vogelgrippe und einem Segment der Hongkong Grippe entstanden. (Ein derartiges Reassortment kann auftreten, wenn die Wirtszelle gleichzeitig mit unterschiedlichen Virustypen infiziert ist.) Abbildung 4A.
Ungewöhnlich erschien auch, daß im Test (Mikroneutralierungs-Test) von Proben junger Menschen unter 24 Jahren die saisonale Grippeimpfung keine Wirkung auf den neuen H1N1-Typ zeigte, bei älteren Populationen über 60 Jahre aber bereits partielle Immunität vorhanden war. Der Grund für diese partielle Immunität konnte damit erklärt werden, daß das für die Immunantwort hauptsächlich verantwortliche Hemagglutinin eine relativ große Ähnlichkeit mit dem Hemagglutinin des 1918-Virus aufweist, das bis in die 1950-Jahre einen großen Teil der älteren Bevölkerung infiziert hatte; diese wiesen offensichtlich noch zirkulierende Antikörper gegen das Virus auf. Der ebenfalls vom 1918-Virus abgeleitete Brisbane H1N1-Subtyp, Grundlage der saisonalen Grippeimpfung 2009, zeigte dagegen eine weitaus größere Veränderung des Hemagglutinins („genetische Distanz“). Die von den Geimpften gegen dieses Hemagglutinin gebildeten Antikörper erkannten das Hemagglutinin der Schweinegrippe nicht und boten somit kaum Schutz vor einer Infektion.
Daß die 2009 Influenza nicht zu einer ähnlichen Katastrophe wie im Jahre 1918 geführt hat, war im wesentlichen darauf zurückzuführen, daß der neue Subtyp das Virulenz Gen (PB1-F2) des 1918-Virus verloren hatte und, daß bereits am Markt vorhandene Inhibitoren der Neuraminidase (Tamiflu, Relenza) hervorragende Wirksamkeit gegen das Virus zeigten. Es erscheint aber durchaus wahrscheinlich, daß zukünftige Pandemien auch unter wesentlich ungünstigeren Bedingungen ablaufen können, wenn ein neuer, hochvirulenter Erreger gegen vorhandene Medikamente resistent ist und wenn nach seiner Identifizierung es noch viele Wochen oder Monate dauert, bis ausreichend Impfstoff verfügbar wird.
Wie hoch beispielsweise das Potential des kürzlich in China aufgetretenen Vogelgrippe-Virus H7N9 (nach Angaben der WHO:131 Erkrankungen und 32 Todesfälle seit Ende März 2013) ist eine Pandemie auszulösen, ist schwer zu prognostizieren. Das Virus wird von Geflügel (in welchem es keine Krankheitssymptome hervorruft) auf den Menschen übertragen; für eine Mensch zu Mensch Übertragung gibt es derzeit keine Bestätigung, diese kann aber durch Mutation des Virus hervorgerufen werden und dann zur schnellen Verbreitung führen. Ebenso besitzt auch das seit mehr als zehn Jahren in einigen Geflügelpopulationen zirkulierende und auf den Menschen übertragbare H5N1-Virus (nach Angaben der WHO von 2003 - 2013: 628 Erkrankungen und 374 Todesfälle) pandemisches Potential. Beide Erregertypen sind sensitiv gegenüber den oben erwähnten Inhibitoren der Neuraminidase.
Um lebensbedrohende Epidemien durch neue, hochinfektiöse Virenstämme abwehren zu können, erscheint die Entwicklung universell gegen unterschiedliche Stämme einsetzbarer, langfristig – möglicherweise sogar lebenslang – wirksamer Impfstoffe vordringlich.
Neue Technologien und Ergebnisse aus der Grundlagenforschung zeigen die Möglichkeit Hemagglutinin-Antikörper zu generieren, die gegen ein sehr breites Spektrum an Influenza Viren immunisieren und damit vor allem auch dem Ausbruch neuer Pandemien entgegenwirken können.
Achillesferse des Hemagglutinins
Hemagglutinin ist derzeit das primäre Zielmolekül (Target) antiviraler Strategien. Gegen dieses, an der Virusoberfläche exprimierte Protein generiert unser Immunsystem die robustesten, neutralisierenden Antikörper als Antwort auf eine natürliche Infektion mit Influenza oder auch auf eine Impfung. Damit wird der primäre Schritt des Anheftens und Eintritts des Virus in die Zelle verhindert:
Das Hemagglutinin-Molekül ragt als pilzförmiger Spike aus der Virusoberfläche heraus. Die Rezeptor-Region, mit der es an die Oberfläche der Wirtszelle andockt, befindet sich in seinem Kopfteil. Rund um diese Region finden sich mehrere Stellen (Epitope), gegen welche Antiköper generiert werden und so aus sterischen Gründen das Anheften an den Wirt verhindern können (Abbildung 5). Derartige – konventionelle – Antikörper sind allerdings nur gegen den speziellen Erregerstamm hochwirksam, da die Epitope im Kopfteil rasch variieren und bereits geringfügige Änderungen in ihrer Aminosäuren-Zusammensetzung (Antigendrift; siehe oben) bewirken können, daß ein Antikörper nicht mehr „paßt“, d.h. unwirksam geworden ist.
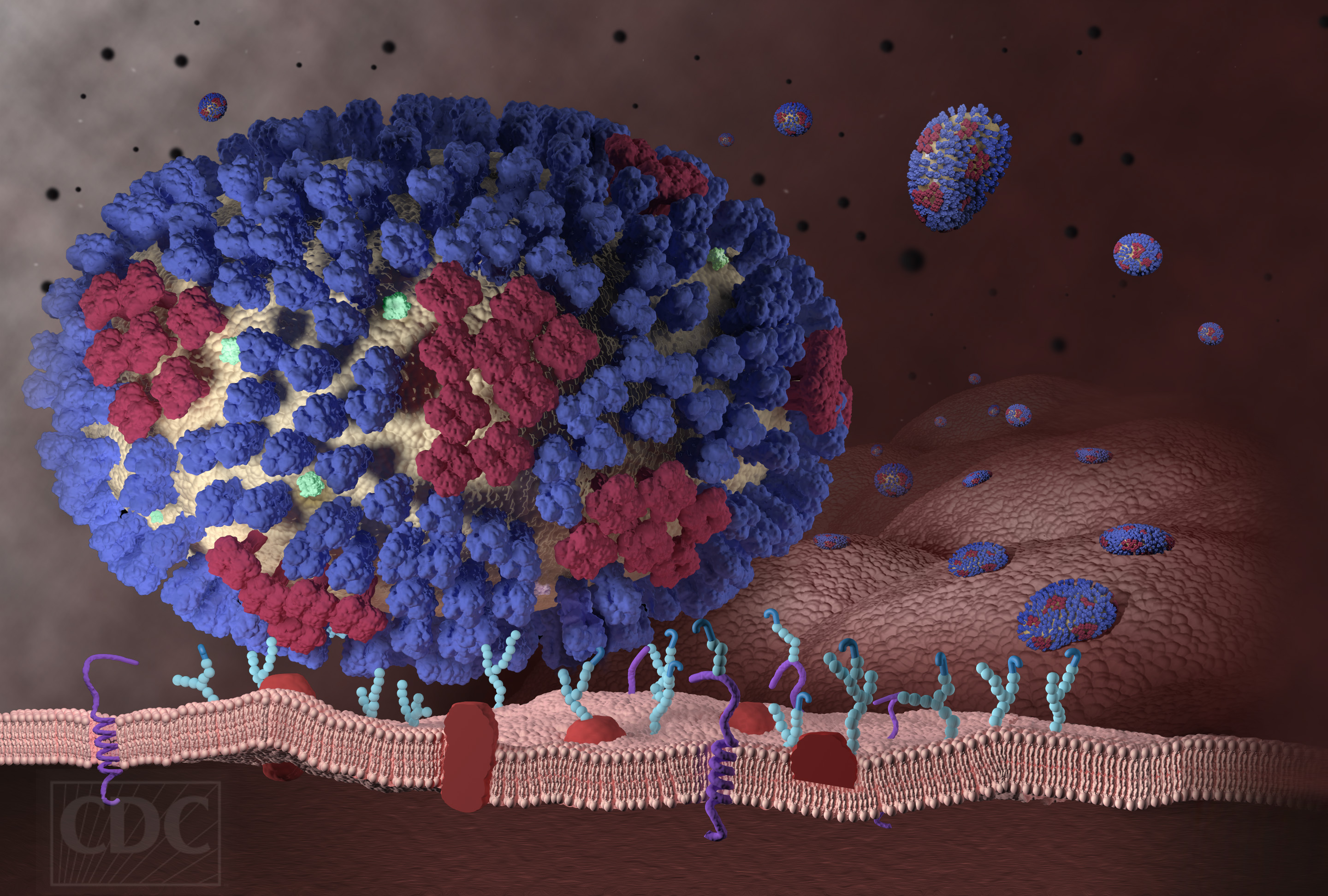
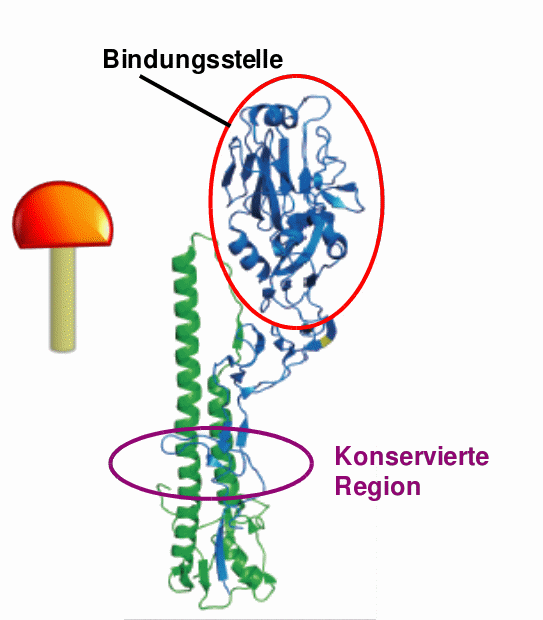 Abbildung 5. Andocken des Influenza A Virus an die Zellmembran einer Wirtszelle. Oben: Bindung des viralen Hemagglutinins (blau) an Kohlehydratreste (hellblau) der Zellmembran des Wirts (rosa). Dunkelrote Spikes an der Virusoberfläche: Neuraminidase. Rechts: „Pilzförmige“ Struktur des Hemagglutinins mit stark variablem Kopfteil (rot eingekreist), an welchem sich die Bindungsstelle für den Wirt befindet, und konservierter Stamm (lila) . (Bilder: http://www.cdc.gov/flu/freeresources/index.htm und Protein databank PDB ID 1RVX)
Abbildung 5. Andocken des Influenza A Virus an die Zellmembran einer Wirtszelle. Oben: Bindung des viralen Hemagglutinins (blau) an Kohlehydratreste (hellblau) der Zellmembran des Wirts (rosa). Dunkelrote Spikes an der Virusoberfläche: Neuraminidase. Rechts: „Pilzförmige“ Struktur des Hemagglutinins mit stark variablem Kopfteil (rot eingekreist), an welchem sich die Bindungsstelle für den Wirt befindet, und konservierter Stamm (lila) . (Bilder: http://www.cdc.gov/flu/freeresources/index.htm und Protein databank PDB ID 1RVX)
Erst vor kurzem wurden beim Menschen nun Antikörper entdeckt, die kreuzreaktiv, d.h. gegen verschiedene Erregertypen, aktiv waren. Untersuchungen ergaben, daß sich diese Antikörper gegen eine, in verschiedensten Virustypen weitgehend konservierte Region des Hemagglutinins richteten, die sich an seinem Stamm befindet (Abbildung 5). In Mäusemodellen konnte gezeigt werden, daß bereits der Stamm – ein synthetisch herstellbares, 60 Aminosäuren langes Peptid – wenn er allein injiziert wird, Immunreaktionen gegen unterschiedliche Erregertypen erzeugte und den andernfalls letalen Ausgang der Infektion verhinderte [2].
Antistamm-Antikörper könnten somit eine gleichermaßen verwundbare Stelle der (meisten) Influenza Viren darstellen. Die im Menschen beobachteten Antistamm-Antikörper bilden langanhaltende Titer, die offensichtlich durch Infektion (beispielsweise mit dem 2009-Virus) und Impfungen noch verstärkt werden. Die Entwicklung von breitest wirksamen Antistamm-Antikörpern und die Möglichkeit deren Effizienz durch Impfungen noch zu verstärken, könnte damit zu neuen Strategien führen, die nicht nur das Problem der saisonalen Virusgrippe, sondern auch das Risiko neuer gefährlicher Pandemien entscheidend reduzieren [3].
[1] WHO recommendations for 2012/2013 influenza season in the northern hemisphere. http://www.euro.who.int/en/what-we-do/health-topics/communicable-disease...
[2] J. Steel et al. (2010) Influenza Virus Vaccine Based on the Conserved Hemagglutinin Stalk Domain. mBio 1(1): doi:10.1128/mBio.00018-10
[3] M.S.Miller et al., (2013) 1976 and 2009 H1N1 Influenza Virus Vaccines Boost Anti-Hemagglutinin Stalk Antibodies in Humans. JID 207:98-105
Weiterführende Links
Zur Wirksamkeit von Grippeimpfungen: siehe https://www.cdc.gov/flu/about/qa/vaccineeffect.htm
European Centre for Disease Prevention and Control (ECDC): Annual epidemiological report 2012. (PDF – free download; 266 p., in English)
Influenza - Die Angriffstaktik des Virus 1:25 min Influenza: Get the (Antigenic) Drift 2:52 min WHO: Clinical evaluation of universal influenza vaccines and pipelines for new influenza vaccines. R.C. Huebner (2013) US Dept. Health & Human Services, ASPR (PDF-slide show, in English).
P.Palese
Pasteur Award 2012 video 1:4 min The Wiley Influenza Virus, Porcine and Otherwise (2009) video 13:49 min. Influenza Pandemics: Past and Future (2006) video 45:12 min The Pathogenicity of Pandemic Influenza Viruses (2008) video 50:58 min
- Printer-friendly version
- Log in to post comments