Fr, 04.04.2014 - 05:20 — Bernhard Rupp ![]()

Vor hundert Jahren schlug mit der Verleihung des Nobelpreises an Max von Laue für seine Entdeckung der Beugung von Röntgenstrahlen an Kristallgittern nicht nur die Geburtsstunde der Kristallographie [1], sondern auch die Geburtsstunde von Max Perutz, einem Pionier der Röntgenkristallographie von Proteinen. Der ursprünglich aus Österreich stammende Chemiker hat – zusammen mit dem Engländer John Kendrew - trotz unüberwindlich scheinender Schwierigkeiten in Jahrzehnte langer Arbeit gezeigt, daß man die Struktur von Proteinen bestimmen kann.
Jeder von uns kennt Kristalle - aus Mineraliensammlungen ebenso wie aus dem Alltagsleben, aus dem die Kristalle kleiner Moleküle wie u.a. von Kochsalz und Zucker nicht wegzudenken sind. Mit Kristallen assoziieren wir üblicherweise Eigenschaften wie Härte, Dauerhaftigkeit und Schönheit.
Auch sehr große Moleküle, selbst die größten Eiweissmoleküle (Proteine), können sich in Form geordneter Kristalle organisieren. Protein-Kristalle sind – wie Abbildung 1 zeigt - nicht weniger schön als die der Mineralien, weisen aber einen entscheidenden Unterschied auf. Sie wachsen zu nur sehr kleinen Kristallen heran und sind sehr empfindlich und zerbrechlich. 
Abbildung 1 Proteinkristalle. Auch große Proteinkristalle sind meistens nur wenige Zehntel Millimeter groß und werden unter dem Mikroskop betrachet. Diese Eigenschaften lassen sich unmittelbar aus den Kristallstrukturen ableiten: i) Die Wechselwirkungen, welche die einzelnen Proteinmoleküle im Kristallgitter zusammenhalten sind schwach im Vergleich zu den Wechselwirkungen zwischen den Atomen im Kristallgitter der Mineralien und ii) der Raum zwischen den Protein-Molekülen – im Mittel an die 50 % - ist mit der Mutterlauge gefüllt, in welche r die Kristalle heranwuchsen (Abbildung 2). Besonders überraschend war es, als die ersten Kristallstrukturen von Proteinen das Fehlen jeglicher Symmetrie in den Molekülen selbst zeigten, diese jedoch schöne symmetrische Kristalle bildeten. 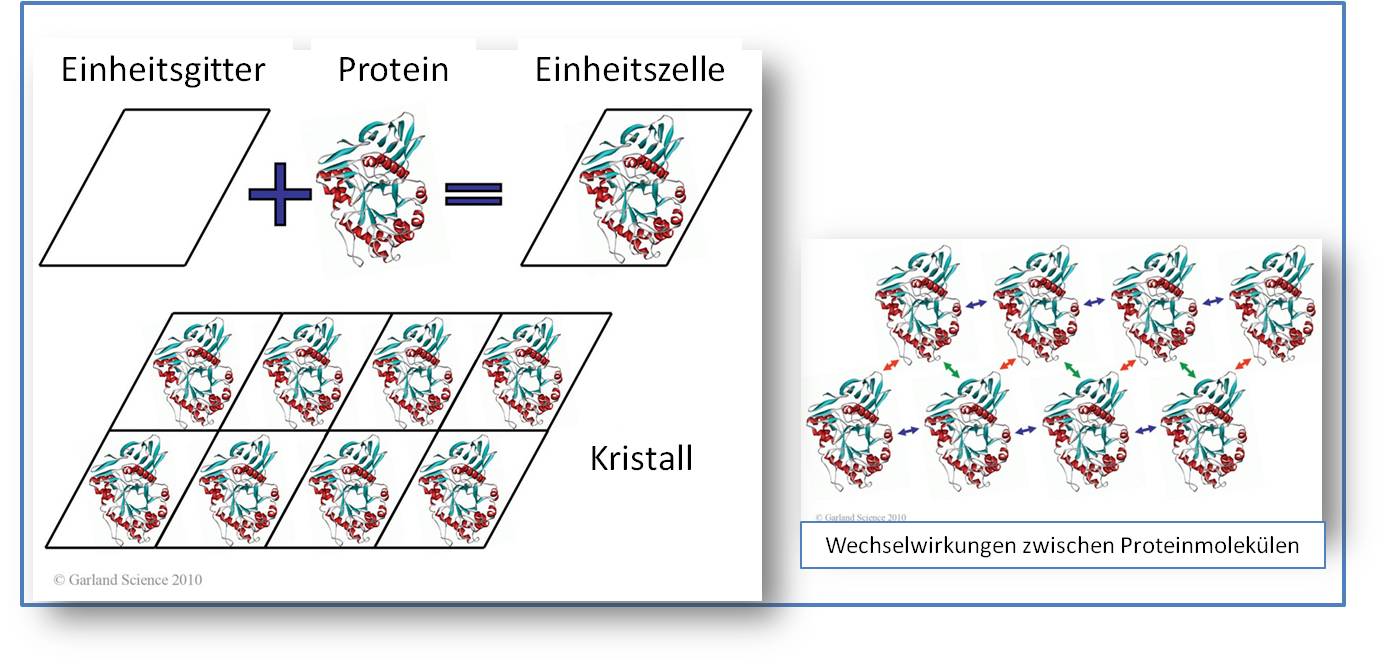 Abbildung 2. Proteinmoleküle im Kristallgitter. Die völlig asymmetrischen Proteinmoleküle organisieren sich zu geordneten Kristallen (vereinfachte zweidimensionale Darstellung). Die intermolekularen Wechselwirkungen zwischen den einzelnen Proteinen (rote, blaue, grüne Pfeile, rechtes Bild) sind relativ schwach. Der verbleibende Raum zwischen den Proteinmolekülen (im Durchschnitt 50 % des Kristallvolumens) ist mit Mutterlauge gefüllt.
Abbildung 2. Proteinmoleküle im Kristallgitter. Die völlig asymmetrischen Proteinmoleküle organisieren sich zu geordneten Kristallen (vereinfachte zweidimensionale Darstellung). Die intermolekularen Wechselwirkungen zwischen den einzelnen Proteinen (rote, blaue, grüne Pfeile, rechtes Bild) sind relativ schwach. Der verbleibende Raum zwischen den Proteinmolekülen (im Durchschnitt 50 % des Kristallvolumens) ist mit Mutterlauge gefüllt.
Wie Proteinkristalle wachsen, kann in einem kurzen Video beobachtet werden [2].
Proteine – von sehr klein bis ganz groß
Proteine sind biologische Makromoleküle mit höchst unterschiedlichem Aufbau, die zahllose, für jedes Leben essentielle Funktionen ausüben:
Proteine regulieren u.a. das Ablesen der in der DNA gespeicherten genetischen Information und deren Übersetzung in Proteine, sie fungieren als Katalysatoren (Enzyme) in Stoffwechselvorgängen, synthetisieren und metabolisieren andere Proteine und bauen Fremdstoffe ab. Proteine, die als Rezeptoren in den Zellmembranen sitzen, ermöglichen den Informationsaustausch zwischen den Zellen und deren Umgebung. Proteine fungieren als Transporter für Moleküle und wiederum andere Proteine bilden die strukturellen Gerüste der Zellen.
Grundbausteine aller Proteine in allen Organismen sind 20 verschiedene Aminosäuren (L-alpha-Aminosäuren), die in unterschiedlicher Reihenfolge (Sequenz) zu linearen Ketten verknüpft (als sogenannte Polypeptide) vorliegen und durch Faltung zu verschiedenartigsten, funktionsspezifischen Strukturen führen. Um dabei räumliche Voraussetzungen zu schaffen, die einzelne biochemische Funktionen ermöglichen, sind Ketten mit mindestens 40 – 50 Aminosäuregruppen notwendig. Von dieser unteren Grenze weg, können Proteine aber auch mehrere tausend Aminosäuregruppen groß werden und dann mehrere funktionelle Domänen enthalten und daher mehrere Funktionen ausüben (Abbildung 3 A, B).
Wenn Proteine mit sich selbst oder mit anderen großen Biomolekülen assoziieren, können noch größere Strukturen entstehen. Beispielsweise treten tausende Aktin-Moleküle (zu je rund 375 Aminosäureresten) zusammen um eine einzige Aktin-Faser zu erzeugen – eine für die Stabilität und Motilität von Zellen, ebenso wie für die Muskelkontraktion essentielle Struktur. Ein weiteres Beispiel sind die Ribosomen – die Maschinerie der Proteinbiosynthese -, riesige Komplexe, die sich aus vielen unterschiedlichen Proteinen und den ribosomalen Ribonukleinsäuren (rRNA‘s) zusammensetzen (Abbildung 3C).
Es ist der Leistungsfähigkeit der Kristallographie, ebenso wie der Ausdauer der Strukturbiologen zu verdanken, daß die Strukturen auch dieser sehr großen Komplexe bis ins atomare Detail bestimmt werden konnten. 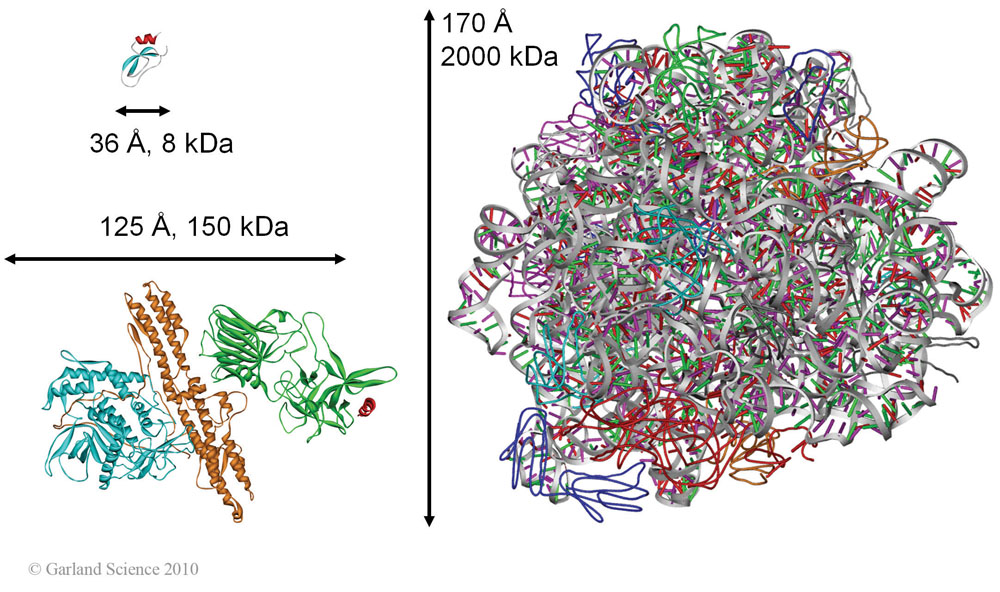 Abbildung 3. Mittels Röntgenkristallographie bestimmte Proteinstrukturen, Beispiele: A) Trypsininhibitor aus Rinderpankreas (PDB 1bpi); ein kleines, aus 58 Aminosäuren bestehendes Protein mit einem Molekulargewicht 8000 Da, notwendig zur Regulierung der Proteinverdauung B) Neurotoxin des Bakteriums Clostridium botulinum (PDB 3bta); die giftigste natürliche Substanz die wir kennen (1 mg tötet ein Pferd): das Protein enthält drei Domänen mit unterschiedlichen Funktionen: (i) Zelladhäsion an Nervenzellen, (ii) Transport in die Nervenzellen, wo (iii) durch die Proteasefunktion der dritten Domäne (links in Abb. B) die Spaltung eines wichtigen intraneuronalen Proteins und in Folge Inaktivierung der Vesikelfusion stattfindet. Die dadurch blockierte Freizsetzung der Neurotransmitter führt zur Muskelparalyse und Erstickung. Darauf beruht auch die kosmetische Wirkung bei subkutaner Injektion: Falten, die durch Anspannung der Gesichtsmuskel entstehen, glätten sich. C) Ribosom – S50 Untereinheit eines Bakteriums (PDB 1ffk); Die 2 000 000 Da große Struktur enthält 27 unterschiedliche Proteine und die ribosomale 5S RNA und 23S RNA. Ribosomen selbst sind die zellulären Maschinen der Proteinproduktion. Alle Strukturen sind als Bändermodelle dargestellt (Längenangaben in Ångstrom (1Å = 0,1 Nanometer), Masse in Dalton (atomare Masseneinheit, 1 Da entspricht der Masse eines Protons)).
Abbildung 3. Mittels Röntgenkristallographie bestimmte Proteinstrukturen, Beispiele: A) Trypsininhibitor aus Rinderpankreas (PDB 1bpi); ein kleines, aus 58 Aminosäuren bestehendes Protein mit einem Molekulargewicht 8000 Da, notwendig zur Regulierung der Proteinverdauung B) Neurotoxin des Bakteriums Clostridium botulinum (PDB 3bta); die giftigste natürliche Substanz die wir kennen (1 mg tötet ein Pferd): das Protein enthält drei Domänen mit unterschiedlichen Funktionen: (i) Zelladhäsion an Nervenzellen, (ii) Transport in die Nervenzellen, wo (iii) durch die Proteasefunktion der dritten Domäne (links in Abb. B) die Spaltung eines wichtigen intraneuronalen Proteins und in Folge Inaktivierung der Vesikelfusion stattfindet. Die dadurch blockierte Freizsetzung der Neurotransmitter führt zur Muskelparalyse und Erstickung. Darauf beruht auch die kosmetische Wirkung bei subkutaner Injektion: Falten, die durch Anspannung der Gesichtsmuskel entstehen, glätten sich. C) Ribosom – S50 Untereinheit eines Bakteriums (PDB 1ffk); Die 2 000 000 Da große Struktur enthält 27 unterschiedliche Proteine und die ribosomale 5S RNA und 23S RNA. Ribosomen selbst sind die zellulären Maschinen der Proteinproduktion. Alle Strukturen sind als Bändermodelle dargestellt (Längenangaben in Ångstrom (1Å = 0,1 Nanometer), Masse in Dalton (atomare Masseneinheit, 1 Da entspricht der Masse eines Protons)).
Von den ersten 3D-Proteinmodellen bis heute
Für die ersten Röntgenstrukturanalysen von Proteinen erhielten der aus Wien stammende Chemiker Max Perutz und der englische Biochemiker John Kendrew den Nobelpreis im Jahre 1962 - fünfzig Jahre nach der Entdeckung der Röntgenstrahlbeugung an Kristallgittern durch Max von Laue [1]. Perutz hatte während seines Studiums an der Universität Wien von faszinierenden biochemischen Untersuchungen in Cambridge gehört und war 1936 für die Erstellung seiner Doktorarbeit dorthin aufgebrochen. 1937 begann er am Cavendish Laboratory die ersten Röntgenbeugungsversuche an Kristallen des Hämoglobin auszuführen, dem relativ kleinen, in den roten Blutkörperchen konzentriert vorliegenden Protein, welches Sauerstoff in der Lunge aufnimmt und im Blutkreislauf an Organe des Körpers wieder abgibt. Zehn Jahre später kam John Kendrew in das Perutz-Labor und begann an der Röntgenstrukturanalyse des Myoglobin zu arbeiten. Dieses, mit Hämoglobin strukturverwandte, aber nur ein Viertel so große Protein dient in den Muskelzellen von Säugetieren der Speicherung und Abgabe von Sauerstoff. Die Versuche von Perutz und Kendrew führten erst in den 1950er Jahren zu ersten brauchbaren Resultaten. Wie die ersten Modelle der Proteinstrukturen aussahen, ist in Abbildung 4 aufgezeigt.  Abbildung 4. Wie Proteinmodelle anfänglich aussahen. Oben links: Myoglobin: dasWurstmodell* von John Kendrew (späte 1950er-Jahre) zeigt nur die Faltung der Aminosäurekette, Oben rechts: Hämoglobin Modell aus Balsaholz von Max Perutz (1959)*. Holzscheiben markieren den Verlauf der Aminosäureketten der 4 Untereinheiten (2 weiße, 2 schwarze). Unten links: John Kendrew und Max Perutz vor dem „Wald von Stäbchen“*, die Kendrew als Basis für die Konstruktion des Myoglobin aus den Aminosäuren benutzte (1960) (Kendrew Modell). Unten rechts: zu restaurierendes Kendrew Modell des Arabinose-Bindungsproteins aus E. coli, welches von Gary Gilliland, Florante Quiocho and George Philips (Rice University) in den frühen 1970-Jahren in den Maßen 1 x 1,5 x 2,5 m erstellt wurde. (* Fotos mit Genehmigung des MRC Laboratory of Molecular Biology).
Abbildung 4. Wie Proteinmodelle anfänglich aussahen. Oben links: Myoglobin: dasWurstmodell* von John Kendrew (späte 1950er-Jahre) zeigt nur die Faltung der Aminosäurekette, Oben rechts: Hämoglobin Modell aus Balsaholz von Max Perutz (1959)*. Holzscheiben markieren den Verlauf der Aminosäureketten der 4 Untereinheiten (2 weiße, 2 schwarze). Unten links: John Kendrew und Max Perutz vor dem „Wald von Stäbchen“*, die Kendrew als Basis für die Konstruktion des Myoglobin aus den Aminosäuren benutzte (1960) (Kendrew Modell). Unten rechts: zu restaurierendes Kendrew Modell des Arabinose-Bindungsproteins aus E. coli, welches von Gary Gilliland, Florante Quiocho and George Philips (Rice University) in den frühen 1970-Jahren in den Maßen 1 x 1,5 x 2,5 m erstellt wurde. (* Fotos mit Genehmigung des MRC Laboratory of Molecular Biology).
In seiner Laudatio anlässlich der Nobelpreisverleihung führte G. Hägg dies so aus: „Als Ergebnis der Arbeiten von Kendrew und Perutz wird es nun möglich die Prinzipien zu sehen, welche dem Aufbau globulärer Proteine zugrundeliegen. Dieses Ziel wurde nach 25 Jahren Arbeit mit anfänglich nur äußerst mäßigen Erfolgen erreicht. Wir bewundern die beiden Wissenschafter daher nicht nur für ihre Genialität und Fertigkeiten, mit denen sie ihre Untersuchungen ausführten, sondern auch für ihre Geduld und Ausdauer, welche die anfangs unüberwindlich scheinenden Schwierigkeiten bewältigten. Wir wissen jetzt, daß man die Struktur von Proteinen bestimmen kann und es ist sicher, daß eine Reihe neuer Bestimmungen bald folgen wird“ [3]
Rund 50 Jahre später sind bereits an die 100 000 Strukturmodelle von Proteinen aus Menschen, Tieren, Pflanzen und Mikroorganismen in der frei zugänglichen Protein Datenbank (PDB) [1, 4] gespeichert. Im Laufe der Jahre wurden die analysierten Strukturen immer größer und komplexer (Abbildung 5). Die Interpretation neuer Beugungsdaten, d.h. die Erstellung von 3D-Struktur-Modellen, erfolgt heute auf Basis all dieser akkumulierten Informationen – ganz zum Unterschied zu den frühen Tagen der Kristallographie, wo die Forscher noch auf keinerlei Basiswissen über Proteinstrukturen zurückgreifen konnten. Auch die Versuchsführung selbst hat sich grundlegend verändert: der gesamte Vorgang von der Kristallisation, Probenahme, Überführung zur Röntgenstrahlungsquelle, raschen Sammlung der Messdaten bis zur hocheffizienten 3D-Strukturbestimmung wurde weitgehend automatisiert und enorm beschleunigt [5]. Die ungeheure Steigerung in Rechenleistung und Speicherfähigkeit moderner Computer ermöglicht, daß auch preiswerte Desktop- oder Laptop-Computer innerhalb von Minuten bis Stunden komplexe kristallographische Berechnungen ausführen können. 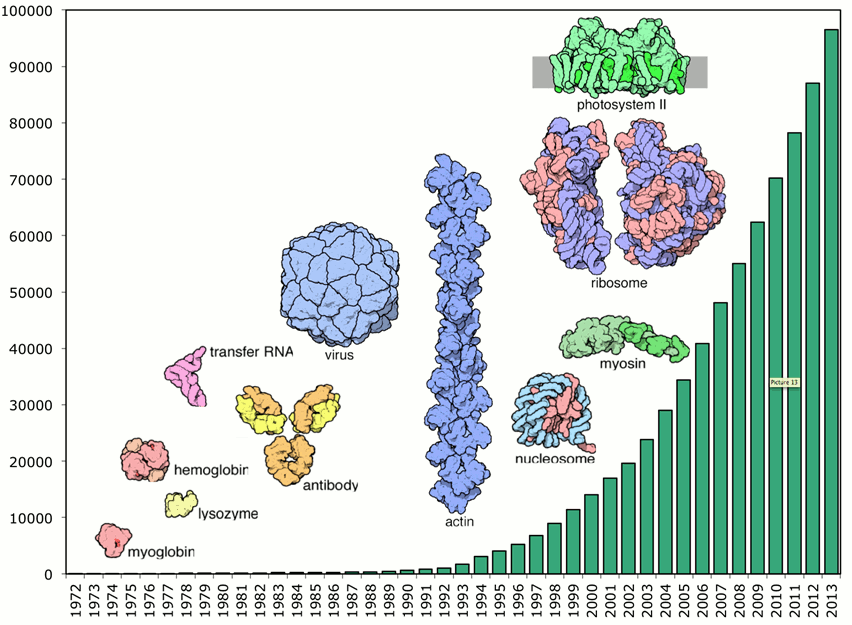 Abbildung 5. Röntgenstrukturanalysen von Proteinen und Proteinkomplexen. Die Zahl an aufgeklärten Strukturen steigt exponentiell an – seit 2008 ist sie auf insgesamt rund 100 000 Strukturen angewachsen [1] - und die untersuchten Moleküle/Molekülkomplexe werden immer größer. Abbildung zusammengestellt von Christine Zardecki, Protein Data Bank (USA).
Abbildung 5. Röntgenstrukturanalysen von Proteinen und Proteinkomplexen. Die Zahl an aufgeklärten Strukturen steigt exponentiell an – seit 2008 ist sie auf insgesamt rund 100 000 Strukturen angewachsen [1] - und die untersuchten Moleküle/Molekülkomplexe werden immer größer. Abbildung zusammengestellt von Christine Zardecki, Protein Data Bank (USA).
Die Möglichkeit Moleküle bis in die atomaren Details betrachten zu können, hat zu einer Revolution im Verstehen der Zusammenhänge von Struktur und Funktion der Proteine geführt und damit die Chance geschaffen, Eigenschaften von Proteinen und damit auch deren Funktionen gezielt zu modulieren. Derartige zielgerichtete Veränderungen sind von grundlegender Bedeutung u.a. in dem Prozess, der zum Auffinden neuer Arzneistoffe führt.
Wie wirken Arzneistoffe?
Um neue Arzneistoffe zu entdecken oder bereits vorhandene entscheidend zu verbessern, werden deren therapeutische Angriffspunkte (Targets) identifiziert - in den meisten Fällen sind dies Proteine, die sich von mit der Krankheit ursächlich verbundenen dysregulierten oder defekten Genen herleiten. Ist ein derartiges Target-Protein gefunden, wird es mittels biotechnologischer Methoden in ausreichender Menge hergestellt (exprimiert), aufgereinigt und nach Möglichkeit zur Kristallisation gebracht. Die Kristallbildung erfolgt häufig vollautomatisch im Hochdurchsatz(High Throughput)-Verfahren, wobei die Kristallisation auch in Gegenwart einer Reihe kleiner Moleküle (Liganden) ausgeführt wird. Aus der Kristallstrukturanalyse des Protein-Liganden Komplexes wird dann ersichtlich, ob und welche Liganden dort andocken und die Funktion des Proteins und damit einen im Zusammenhang stehenden Krankheitsprozess maßgeblich beeinflussen könnten. Derartige Moleküle dienen dann als Leitverbindungen – sogenannte „Leads“ - für die Entwicklung neuer, spezifischer Arzneistoffe. Der Prozess ist in Abbildung 6 vereinfacht dargestellt. 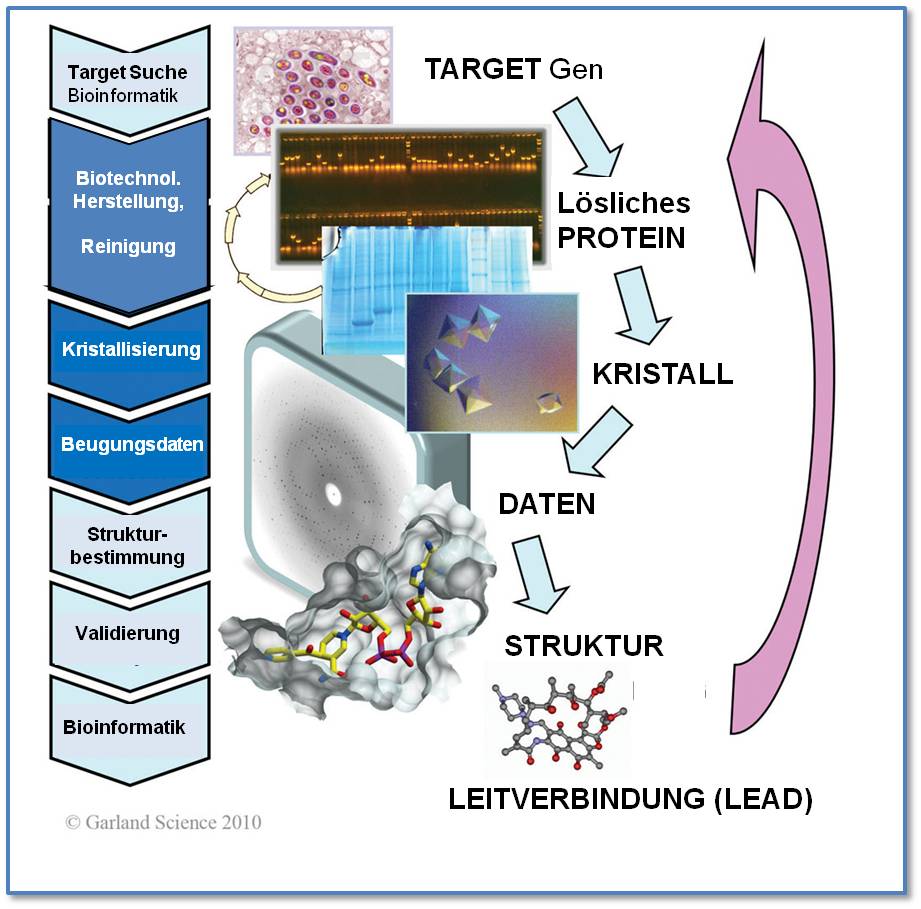 Abbildung 6. Entdeckung neuer Wirkstoffe. Unterschiede in der Genexpression von krankem (z. B Krebs) versus gesundem Gewebe (Differential Genomics) weisen auf falsch regulierte und/oder defekte Gene hin: deren Genprodukte – Proteine – können geeignete Targets für die Auffindung neuer Arzneistoffe sein und werden, wie im Text beschrieben, untersucht. Als Beispiel der röntgenkristallographischen Untersuchungen an bakteriellen Zielproteinen ist im unteren Teil des Bildes dargestellt, wie die aktive Form des gegen Tuberkulose angewandten Standard-Medikaments Isoniazid (als Stäbchenmodell) in einer Bindungstasche des bakteriellen Target-Enzyms positioniert ist, darunter die Struktur eines Moleküls, das als Leitverbindung für die Synthese eines neuen, effizienteren Arzneistoffes gegen Antibiotika-resistente Bakterien dient.
Abbildung 6. Entdeckung neuer Wirkstoffe. Unterschiede in der Genexpression von krankem (z. B Krebs) versus gesundem Gewebe (Differential Genomics) weisen auf falsch regulierte und/oder defekte Gene hin: deren Genprodukte – Proteine – können geeignete Targets für die Auffindung neuer Arzneistoffe sein und werden, wie im Text beschrieben, untersucht. Als Beispiel der röntgenkristallographischen Untersuchungen an bakteriellen Zielproteinen ist im unteren Teil des Bildes dargestellt, wie die aktive Form des gegen Tuberkulose angewandten Standard-Medikaments Isoniazid (als Stäbchenmodell) in einer Bindungstasche des bakteriellen Target-Enzyms positioniert ist, darunter die Struktur eines Moleküls, das als Leitverbindung für die Synthese eines neuen, effizienteren Arzneistoffes gegen Antibiotika-resistente Bakterien dient.
Die Röntgenkristallographie zeigt auf, wie und warum kleine Moleküle etwa bakterielle Proteine blockieren, die essentiell mit der Funktion eines Krankheitserregers verknüpft sind, oder wie sie beispielsweise als Inhibitoren von Rezeptorproteinen fungieren, welche Tumorzellen für ihr unkontrolliertes Wachstum benötigen. Darüber hinaus erlaubt es die genaue Kenntnis der Geometrie und der elektrochemischen Eigenschaften des Bindungsortes Strukturen zu designen, die sich an die Gegebenheiten optimal anpassen und damit zu höherer therapeutischer Wirksamkeit führen sollten.
Als Beispiel sei hier Isoniazid angeführt, ein bereits seit langem verwendetes Standard-Medikament gegen Mykobakterien, die Erreger der Tuberkulose. Dieses sehr kleine (synthetische) Molekül wird erst im Bakterium in eine aktive Form (isonicotinic acyl-NADH) überführt, die dann an ein Enzym bindet, das für die Synthese bestimmter langkettiger Fettsäuren (Mykolsäuren) in der bakteriellen Zellwand verantwortlich ist. Die Kristallographie demonstriert, wie diese aktive Form in der Bindungstasche des Enzyms positioniert ist, bestätigt damit den Wirkungsmechanismus und zeigt Lead-Strukturen zur Optimierung der Bindung und Wirksamkeit auf (Abbildung 5, unten). Zum Glück haben menschliche Zellen nicht die gleichen Fettsäure-reichen Zellwände wie die Mykobakterien, und wir benötigen daher auch kein Enzym für die Synthese der Mykolsäuren. Der Inhibitor Isoniazid wird daher von Patienten weitgehend problemlos vertragen, während die infektiösen Mykobakterien absterben. Aufkommender Resistenzentwicklung auf Grund von Mutationen in der Proteinstruktur kann in vielen Fällen mit der Strukturanalyse des veränderten Proteins und der Anpassung des Wirkstoffes an die neuen Voraussetzungen begegnet werden.
Fazit
In den nun 100 Jahren ihres Bestehens hat die Röntgenkristallographie enorm zum Fortschritt aller Disziplinen von Wissenschaft und Technologie beigetragen, die sich mit kristallisierbaren Materialien beschäftigen. Auf der Basis der bis in die atomaren Details analysierten Kristallstrukturen werden die Eigenschaften von Materialien verstanden, können verbessert oder auch neu geplant werden, ob es nun Materialien aus der anorganischen Welt sind – wie beispielsweise Halbleiter, Legierungen und Stoffe, die in der Raumfahrt verwendet werden. In den Biowissenschaften ermöglicht die Kristallographie den rasanten Fortschritt in unseren Kenntnissen zu Aufbau und Wirkungsmechanismen von Biomolekülen und schafft die Basis gezielt effiziente Wirkstoffe gegen Krankheiten zu entwickeln.
[1] Bernhard Rupp, Wunderwelt der Kristalle — Die Kristallographie feiert ihren 100. Geburtstag
[2] Proteinkristallen beim Wachsen zusehen: Video von George Sheldrick and Students (Göttingen) http://www.ruppweb.org/iycr/low.m1v (low resolution, 1.8MB);
http://www.ruppweb.org/iycr/high.m1v (high resolution, 5MB)
[3] Award Ceremony Speech, Presentation Speech by Professor G. Hägg, member of the Nobel Committee for Chemistry of the Royal Swedish Academy of Sciences.
http://www.nobelprize.org/nobel_prizes/chemistry/laureates/1962/press.html
[4] Protein Data Bank http://www.wwpdb.org/stats.html
[5] Von den Kristallen bis zur 3D-Struktur eines Proteins (Phosphodiesterase). Video (ca 4 min; Englisch). www.ruppweb.org/cryscam/drugs_and_bots_small.wmv
Weiterführende Links
- Homepage von Bernhard Rupp: http://www.ruppweb.org/iycr/IYCr_2014.htm
- Bernhard Rupp: Biomolecular Crystallography: Principles, Practice, and Application to Structural Biology (2009): das umfassende Lehrbuch über Grundlagen, Techniken und Anwendungen der Kristallographie in der Strukturbiologie. http://www.ruppweb.org/garland/default.htm
- Internationales Jahr der Kristallographie: http://www.iycr2014.de /
- Max Perutz: X-ray analysis of haemoglobin, Nobel Lecture, December 11, 1962; http://research.chem.psu.edu/sasgroup/chem540/downloads/perutz-lecture.pdf
- Gottfried Schatz: Porträt eines Proteins. — Die Komplexität lebender Materie als Vermittlerin zwischen Wissenschaft und Kunst.
- What is a Protein? http://www.rcsb.org/pdb/education_discussion/educational_resources/what_...
- Video: Celebrating Crystallography 3:05 min. (Englisch) http://www.richannel.org/celebrating-crystallography
- Video: Introduction to X-ray crystallography 17.26 min (harvardbmw’s videos; Englisch)) http://vimeo.com/7643687
- Video: Myoglobin - A brief history of structural biology. 4,37 min (Englisch) http://www.richannel.org/collections/2013/crystallography#/myoglobin-a-b...
- Video: A case of crystal clarity http://www.richannel.org/collections/2013/crystallography#/a-case-of-cry... 2,37 min. (Englisch)
- Printer-friendly version
- Log in to post comments
