Do, 13.09.2012- 00:00 — Peter Schuster
Zentralismus versagt in der Kontrolle hochkomplexer Systeme. Ein eindrucksvolles Beispiel aus den Regulationsmechanismen der Natur. Die Ineffizienz zentraler Kontrollen ist uns allen aus unserem täglichen Leben bekannt. In der Natur ist das Problem der Regulation komplexer Systeme, wie der Genexpression in höheren Organismen, durch ein Zusammenspiel von zentraler und dezentralisierter Kontrolle gelöst.
Die Aussage, daß eine zentrale Kontrolle großer, komplexer Einheiten zum Scheitern verurteilt ist, stellt eine Binsenweisheit dar. Wirtschaft und Gesellschaft untermauern die Gültigkeit dieser Aussage durch zahllose Beispiele in der Vergangenheit und Gegenwart, welche beweisen: Systeme werden ineffizient, sobald sie eine kritische Größe überschreiten. Bereits in frühen Zeiten waren sich kluge Herrscher dieses Problems bewußt und haben dem eine Strategie des „divide et impera“ entgegengesetzt.
Die Natur scheint eine elegante Lösung für die Regulation ihrer großen, hochkomplexen Systeme gefunden zu haben, indem sie modulare Strukturen verwendet und die Module darin partielle Autonomie besitzen. Das beste Beispiel dafür ist der Vielzeller-Organismus, in welchem jede einzelne Zelle über gerade so viel Autonomie in ihrem Stoffwechsel verfügt, als toleriert werden kann ohne dem gesamten System Schaden zuzufügen: Ein wenig mehr Unabhängigkeit für somatische Zellen und unlimitiertes Wachstum – ein Tumor – kann entstehen.
Zentrale Kontrolle in Bakterien
Die Regulierung von Stoffwechsel und Vermehrung der einfachsten Lebensformen, der Bakterien, wird noch zentral gesteuert. Um möglichst effizient zu bleiben, sich möglichst effizient zu vermehren, ist den Bakterien in der Größe ihres Genoms aber eine Obergrenze gesetzt. Wie aus der Sequenzierung von mehr als tausend bakteriellen Genomen hervorgeht, ist es die darin enthaltene Zahl an regulatorischen Genen, welche die Größe des Genoms limitiert [1, 2]: Bakterien mit einem kleinen Genom von einigen hundert Genen besitzen nur wenige dieser regulatorischen Gene. Mit wachsender Größe des Genoms nimmt deren Zahl aber in einer quadratischen Funktion zu (Abbildung 1) und führt schließlich dazu, daß jedes der nicht-regulatorischen Gene – welche ja für die in Stoffwechsel und Vermehrung essentiellen Proteine kodieren - ein eigenes regulatorisches Gen zur Steuerung besitzt. Es ist evident, daß eine weitere Steigerung dieses „Überwachungs-/Kontrollsystems“ zu „kostspielig“ wird, eine maximale Größe erreicht ist.
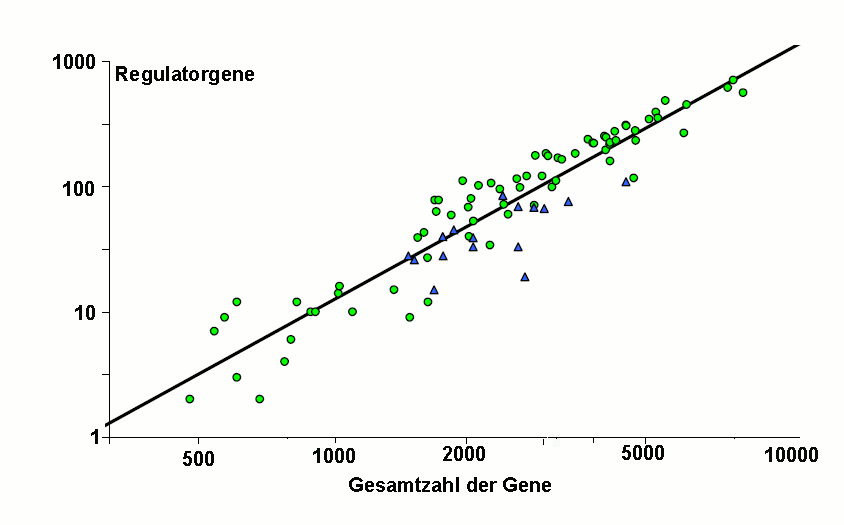 Abbildung 1. Die Zahl der regulatorischen Gene im bakteriellen Genom wächst mit dessen Größe im quadratischen Ausmaß. Die Zahl der Gene im Genom und die Zahl der Regulatorgene sind im logarithmischen Maßstab dargestellt. Meßwerte in Grün stammen von Archäbakterien, in Blau von Bakterien. Bild modifiziert nach Croft et al., [2].
Abbildung 1. Die Zahl der regulatorischen Gene im bakteriellen Genom wächst mit dessen Größe im quadratischen Ausmaß. Die Zahl der Gene im Genom und die Zahl der Regulatorgene sind im logarithmischen Maßstab dargestellt. Meßwerte in Grün stammen von Archäbakterien, in Blau von Bakterien. Bild modifiziert nach Croft et al., [2].
Zusammenspiel von zentraler Kontrolle und dezentralisierter Regulation in höheren Organismen
Eukaryonten – das sind alle höheren Organismen von der Hefe bis zum Menschen – benutzen für die Regulation ihrer wesentlich komplexeren Systeme der Genexpression ein Zusammenspiel von zentraler Kontrolle und dezentralisierter Regulation:
- Zentral ist dabei die genetische Kontrolle auf der DNA-Ebene, d.h. auf der Ebene der Übersetzung der in den Genen festgelegten, noch rohen Baupläne für Proteine.
- Dezentralisiert erfolgt die Regulation der in die Vorläufer-Messenger RNA (pre-mRNA) kopierten (transkribierten) Baupläne durch alternatives Auseinanderschneiden (splicing) und Zusammenfügen von Abschnitten der pre-mRNA zu reifen Messenger-RNAs, den für die Synthese der Proteine verwendeten Vorlagen (siehe unten, Abbildung 2) und
- Eine Reihe von epigenetischen Regulationsmechanismen greifen am Gen selbst oder in späteren Phasen der Übersetzung regulierend ein.
Die Erforschung des Zusammenwirkens dieser Mechanismen ist neuesten Datums, basierend auf der im Human Genome (HUGO) Projekt erfolgten Entschlüsselung des menschlichen Genoms, d.h. aller Gene, welche die Baupläne für die den Aufbau und Stoffwechsel unseres Organismus bestimmenden Proteine enthalten. Damals, im Jahr 2001, war die Verwunderung groß, als beim Menschen mit rund 22 000 für Proteine kodierenden Genen nicht viel mehr Gene als beispielsweise beim Wurm Caenorhabditis (rund 19000 Gene) festgestellt wurden und diese insgesamt nur rund 1,5 % des gesamten Genoms ausmachten.
Das ENCODE-Projekt
Welche Funktion haben nun die restlichen – nicht für Proteine kodierenden – 98,5 % der DNA, sind diese nicht – wie häufig angenommen - zum Großteil nur Müll (junk DNA)? Ein riesiges internationales Team von 442 Forschern hat sich mit dem „Encyclopedia of DNA Elements (ENCODE)-Projekt“ die systematische Erkundung aller funktionellen Elemente der DNA zum Ziel gesetzt und vor wenigen Tagen die ersten Ergebnisse in rund 30, öffentlich zugänglichen Publikationen veröffentlicht [2]. Demnach konnten bereits zu rund 80 % der DNA biochemische Funktionen zugeordnet werden, die direkt – zentral - am Chromosom oder indirekt über RNA-Transkripte, die nicht in Proteine übersetzt werden und über weitere Mechanismen regulieren wann, in welchen Zelltypen, unter welchen Bedingungen und in welchem Ausmaß Gene abgelesen und in Proteine übersetzt werden.
Für unsere Frage nach zentraler und dezentraler Regulierung kommt dabei besondere Bedeutung dem Mechanismus zu, mit dem die ursprünglich in der genetischen Information vorhandenen Baupläne in funktionelle Proteine übersetzt werden, dem Vorgang des alternativen Splicing der pre-mRNA (siehe oben).
Hier wendet die Natur erfolgreich ein Modul-Schema an (Abbildung 2): Dasselbe Stück DNA, das von einem Gen in eine pre-mRNA transkribiert wird, kann durch unterschiedliches Herausschneiden und Zusammenfügen diverser Abschnitte zur Bildung mehrerer reifer mRNA Moleküle und in der Folge zu unterschiedlichen Proteinen führen. Wie ENCODE zeigt, folgt diese Expression keiner minimalistischen Strategie: abhängig vom individuellen Organismus und den Zelltypen/Geweben in welchen die Expression stattfindet, entstehen aus einer pre-mRNA simultan bis zu 10 – 12 Isoformen, wobei jeweils zumindest zwei Isoformen vorherrschen.
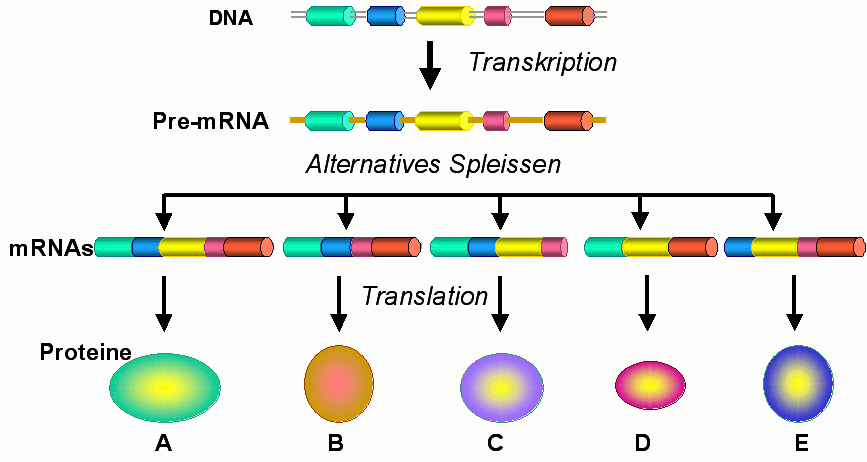 Abbildung 2. Genexpression und Alternatives Spleißen in eukaryontischen Zellen: Aus ein und demselben Gen, das in eine pre-mRNA kopiert (transkribiert) wurde, entstehen durch Alternatives Spleißen dieser pre-mRNA mehrere unterschiedliche reife mRNAs und aus diesen durch Proteinsynthese an den Ribosomen (Translation) eine Reihe verschiedener Protein-Isoformen.
Abbildung 2. Genexpression und Alternatives Spleißen in eukaryontischen Zellen: Aus ein und demselben Gen, das in eine pre-mRNA kopiert (transkribiert) wurde, entstehen durch Alternatives Spleißen dieser pre-mRNA mehrere unterschiedliche reife mRNAs und aus diesen durch Proteinsynthese an den Ribosomen (Translation) eine Reihe verschiedener Protein-Isoformen.
Zweifellos ist das von ENCODE gezeichnete Bild noch ein sehr unvollständiges, und wir müssen auf Überraschungen gefasst sein. Was aber als gesichert angesehen werden kann, ist, daß es Eukaryonten gelungen ist, durch dezentrale Regulierungsmechanismen – einem „divide et impera“ - die Limitierung in der Anzahl ihrer Gene aufzuheben und höchstkomplexe Systeme zu steuern.
Zentrale – dezentrale Kontrolle: Versuch einer ökonomischen Betrachtung
Um eine grobe Abschätzung der unterschiedlichen „Kosten“ von zentraler versus dezentraler Kontrolle zu treffen, wollen wir ein zentral reguliertes, 10 000 Gene enthaltendes Genom eines Bakteriums vergleichen mit einem gleich großen, virtuellen System, das aber aus 10 lokal regulierten Genomen zu je 1000 Genen bestehen soll:
Im Fall der zentralen Organisation werden – wie aus Abbildung 1 ableitbar - rund 1200 Regulatorgene benötigt [1, 2]. Im dezentralen System reicht dagegen ein kleines Zentrum - beispielsweise mit 180 Regulatorgenen (jeweils 4 Gene für die insgesamt 45 paarweisen Wechselwirkungen) – aus um die Aktivitäten der 10 Untersysteme kontrollieren, je 12 Gene pro Untersystem sollten für dessen Regulation genügen und jeweils 2 „Kontrolloren“ für die Meldungen aus den Untersystemen an das Zentrum. In Summe könnte damit das skizzierte dezentralisierte Modell mit insgesamt 320 Regulatorgenen - einem Viertel des Bedarfs des zentralen Modells - sein Auslangen finden.
Fairerweise muß man dazu anmerken, daß bei einem Einsparen von rund ¾ der Kontrolloren im dezentralisierten Modell auch einiges an Information verloren gegangen ist. Der Großteil der Kontrollfunktionen wird lokal geregelt, das „Nachrichten-Hauptquartier“ erhält gerade soviel an Information, wie für das Management der Wechselwirkungen zwischen den Untersystemen absolut erforderlich ist und kann damit kaum Planungen für die Zukunft des gesamten Systems anstellen. Derartige Konzepte können ja kaum ohne ein umfassendes Bild des gesamten Umfeldes und der daraus abgeleiteten Schlußfolgerungen erstellt werden.
Der Darwin`sche Mechanismus einer Optimierung durch Variation und Selektion braucht sich aber um die Zukunft nicht zu kümmern, da er ja nach einem Prinzip von „trial and error“ vorgeht. Für sich allein existierende Vielzeller-Organismen können damit leicht mit einem Mindestmaß an zentraler Kontrolle und „Zukunftsplanung“ auskommen. Natürlich ändert sich die Situation, wenn es um Gesellschaften im Tier- und Menschenreich geht. Erziehung und Lernen des einzelnen Individuums verändern hier den Darwinischen Mechanismus in einen Lamarckschen: Information, die ein einzelnes Mitglied der Gesellschaft erhalten hat, kann schnell und weitgehend auch an zukünftige Generationen weitergegeben werden, zentralisierte Prognosen über zukünftige Entwicklungen liegen daher im Interesse der Gesellschaft. Ein Zuviel an Föderalismus, ein Zuwenig an zentraler Steuerung ermöglichen hier kaum eine erfolgreiche Planung für die Zukunft. Dazu möchte ich nur ein zur Zeit wichtiges Beispiel anführen: das Versagen einer weltweiten Reduktion der Kohlendioxyd-Emissionen trotz eindeutigen Nachweises von dessen Notwendigkeit.
Schlußfolgerung aus akademischer Sicht
Die Ineffizienz zentraler Kontrollen kennen wir alle aus unserem täglichen Leben. Wenn man an einer großen Universität mit mehr als 80 000 Studenten arbeitet, möchte man die Lehre aus dem Beispiel der Bakterien ziehen, daß die Größe eines Systems limitiert ist, das zentral verwaltet werden kann. Für eine dezentralisierte Universitätsverwaltung sprechen viele Gründe, einer davon liegt darin die Kosten für die „Overheads“ gering zu halten, da diese ja die ohnehin stark begrenzten Mittel der Forschungsgelder dramatisch reduzieren:
Wir sollten daher auf unsere Forschungstätigkeit fokussiert sein und nicht darauf unsere Dekane und Rektoren permanent über Ideen und Planungen zu unterrichten, die sich möglicherweise nicht realisieren lassen, wenn wir - wie es ja unsere Aufgabe ist - ins Neuland vorstoßen. Der Erfolg dieses Vorgehens läßt sich an den Veröffentlichungen in der wissenschaftlichen Literatur sehr gut ablesen und bietet damit die Basis für Evaluierungen durch die zentrale Verwaltung. In den meisten anderen Belangen sollte sich diese aber heraushalten und kann damit so klein wie möglich gehalten werden.
Einzelnachweise und Quellen:
[1] EV Koonin, YI Wolf (2008) Genomics of bacteria and archaea: the emerging dynamic view of the prokaryotic world. Nucleic Acids Res. 36:6688-6719.
[2] Croft, L.J.; Lercher, M.J.; Gagen, M.J.; Mattick, J.S.(2003) Is prokaryotic complexity limited by accelerated growth in regulatory overhead? Genome Biology, 5:P2
[3] The ENCODE Project Consortium. An integrated encyclopedia of DNA elements in the human genome. Nature 489: 57- 74. 6 September 2012
Glossar
DNA: Desoxyribonucleinsäure; ein üblicherweise in Form eines Doppelstranges (Doppelhelix) vorliegendes Polymer, welches die primäre genetische Information aller Zellen enthält.
RNA: Ribonucleinsäure. Polymere, welche unterschiedliche biochemische Funktionen ausüben können, in der Umsetzung von genetischer Information (Genexpression) ebenso wie als katalytisch aktive Moleküle.
Pre-mRNA: primäres Transkript (Umschreibung) eines zu einem Gen gehörenden Abschnitts der DNA in ein mRNA-Molekül, durch alternatives Splicen entsteht daraus die messenger-RNA (mRNA).
Alternatives Splicen: Abschnitte der pre-mRNA, die keine kodierende Informationen enthalten (Introns), werden entfernt und die verbleibenden Abschnitte (Exons) in unterschiedlicher Weise miteinander verbunden. Damit können aus ein und demselben Gen/derselben pre-mRNA mehrere verschiedene reife mRNA-Moleküle entstehen und aus diesen mehrere unterschiedliche Proteine.
Weiterführende Links
ENCODE: Encyclopedia Of DNA Elements (video 4,4 min) ENCODE, the Encyclopaedia of DNA Elements, is the most ambitious human genetics project to date. ENCODE: Encyclopedia of DNA Elements (video 6,5 min) Ewan Birney of EMBL-EBI, Tim Hubbard of the Wellcome Trust Sanger Institute and Roderic Guigo of CRG talk about ENCODE, an international project which revealed that much of what has been called 'junk DNA' in the human genome is actually a massive control panel with millions of switches regulating the activity of our genes.
Read more about this at: http://www.embl.org/press/2012/120905_Hinxton.
- Printer-friendly version
- Log in to post comments

